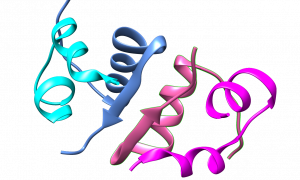
Das rätselhafte Protein: Eine Bioinformatik-Expedition
Übersicht
Gemeinsam werden wir in die faszinierende Welt der Bioinformatik eintauchen, um die Geheimnisse von Proteinen zu entschlüsseln, einem der grundlegenden Bausteine der Molekularbiologie. Anhand einer Reihe von interaktiven Übungen werden wir untersuchen, wie Proteine hergestellt werden, wie sie aussehen und wie sie sich über die verschiedenen Arten hinweg entwickelt haben. Begleite uns auf diesem fiktiven wissenschaftlichen Abenteuer, das mit realen Forschungsaktivitäten am EMBL verwoben ist, und gewinne dabei Einblicke in moderne Forschungsmethoden.
Ziel dieser Aktivität ist es, unser Verständnis von Proteinen und ihrer Evolution zu vertiefen und gleichzeitig wertvolle Einblicke in die praktischen Anwendungen der Bioinformatik in der wissenschaftlichen Forschung zu geben.
Deine Aufgabe
Während der TREC-Expedition (Traversing European Coastlines) sammeln Forscher*innen zahlreiche biologische Proben von der europäischen Küste, um diese Ökosysteme zu untersuchen. Diese Proben werden aus dem Boden, Sedimenten, Aerosol, Flachwasser und dem Meer gewonnen und mit wissenschaftlichen Techniken wie Mikroskopie oder DNA-Sequenzierung sorgfältig analysiert.
Kürzlich stieß das TREC-Team auf eine verblüffende Entdeckung in einer ihrer Proben aus dem flachen Wasser – eine DNA-Sequenz, die mit keiner bekannten Sequenz übereinstimmt. Die Forscher*innen sind von diesem Fund fasziniert und haben eine Aufgabe für dich: Kannst du ihnen helfen, das von dieser Sequenz kodierte Protein zu identifizieren und die biologische Funktion des Proteins zu entschlüsseln? Kannst du außerdem dabei helfen, die Spezies zu identifizieren, von der die Sequenz stammt und die evolutionären Beziehungen zu anderen Organismen zu erforschen?
Bioinformatische Methoden
Die meisten der Methoden, die wir im Rahmen dieser Aktivität einsetzen werden, wurden vom EMBL-EBI entwickelt und Wissenschaftler*innen in aller Welt kostenlos zur Verfügung gestellt.
EMBOSS Transeq
Link: https://www.ebi.ac.uk/Tools/st/emboss_transeq/
Eine Methode zur Übersetzung von Nukleinsäuresequenzen (DNA oder RNA) in ihre entsprechenden Aminosäuresequenzen.
NCBI BLAST+ (Basic Local Alignment Search Tool)
Link: http://www.ebi.ac.uk/Tools/sss/ncbiblast/
Eine Methode zur Identifizierung und zum Vergleich biologischer Sequenzinformationen, wie z. B. Aminosäuresequenzen. Es ermöglicht Forscher*innen ein bestimmtes Protein mit einer Datenbank von Sequenzen zu vergleichen. Dies ermöglicht die Identifizierung unbekannter Proteine.
Clustal Omega
Link: https://www.ebi.ac.uk/Tools/msa/clustalo/
Eine Methode für das Alignment und den Vergleich mehrerer Sequenzen, die speziell für Aminosäuresequenzen geeignet ist.
Simple Phylogeny
Link: https://www.ebi.ac.uk/Tools/phylogeny/simple_phylogeny/
Eine Methode zur Durchführung grundlegender phylogenetischer Analysen an einem multiplen Sequenzalignment.
UniProt-Datenbank (Universal Protein Resource)
Link: http://www.uniprot.org/
Ein umfassender Katalog von Proteininformationen, einschließlich Proteinsequenzen und deren Funktionen.
AlphaFold Datenbank
Link: https://alphafold.ebi.ac.uk/
Eine Datenbank für Proteinstrukturvorhersagen. AlphaFold ist ein künstliches Intelligenz (KI) System, das die 3D-Struktur eines Proteins auf der Grundlage seiner Aminosäuresequenz vorhersagen kann. Die AlphaFold-Datenbank ist auch in die UniProt-Datenbank integriert.
Hinweis: Die Methoden benötigen einige Zeit für die Berechnungen. Daher kann ein wenig Geduld erforderlich sein.
Aktivitäten-Navigation
● Teil 1: Von der DNA zur Proteinsequenz
● Teil 2: Identität und Funktion von Proteinen
● Teil 3: Multiples Sequenzalignment
Topic area: Bioinformatics, Evolutionary biology, Structural & Computational biology, Biochemistry
Type of resource: Online resource
Age group: 16-19
Contact: SEPE team
Author: SEPE
Share:
 English
English